差异基因与细胞标注
在单细胞RNA测序分析中,聚类之后筛选差异基因的主要目的是为了深入理解不同细胞群体之间的生物学差异。首先先看我们筛选出来的数据并对其进行解释
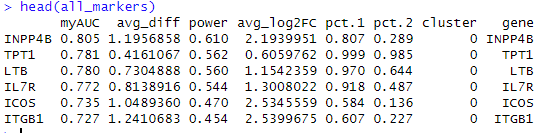
**p_val:**基因表达量差异P值(一般不看这个)
**p_val_adj:**校正后的P值(一般看这个)
**avg_log2FC:**基因在该细胞簇中与其他细�胞簇表达量差异倍数的log值,一般大于2是最好的效果,说明差异很大
**pct.1:**在该细胞簇中表达该基因的细胞数量占比
**pct.2:**在其他细胞簇中表达该基因的细胞数量占比平均值
**cluster:**在哪一类簇中
**gene:**名字
**myroc:**roc评分,范围从[0,1] ,越大越好
我们做差异基因检查的目的是什么呢?
- 验证聚类结果
聚类过程将细胞分为不同的群体,但这些群体是否具有生物学意义需要进一步验证。筛选出差异基因可以帮助确定这些聚类是否合理。并且,如果在不同的聚类之间找到显著差异的基因,说明这些群体之间可能存在生物学差异,证明聚类是合理的。
- 标记细胞群体
放在后文讲,这就是细胞标注
- 下游分析
这些基因是下游基因的基础(GO分析,KEGG,拟时序,cell-cell communication等)
- 优化聚类
通过筛选差异基因,可以对初步聚类结果进行评估
如何对差异基因进行评估?首先,前者的roc评分可以作为一个依据,其次,可以通过可视化进行观察
该基因在其他簇中的分布情况(小提琴图与聚类特征标注图)
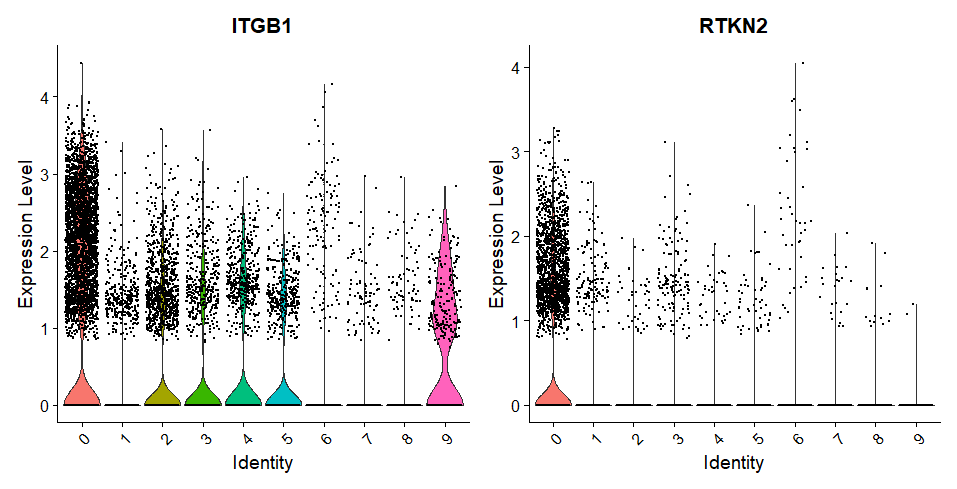
通过小提琴图可以观测出,ITBG1这个基因其实在每个簇中都有,差异基因是通过比较其他簇得出的,这就说明其实这个基因的差异性不是很明显,相反RTKN2更有差异性,我们做到差异基因这一步的时候,必须要有该簇的分布与其他簇不同的结果,这样后续细胞标记出的结果也会更好
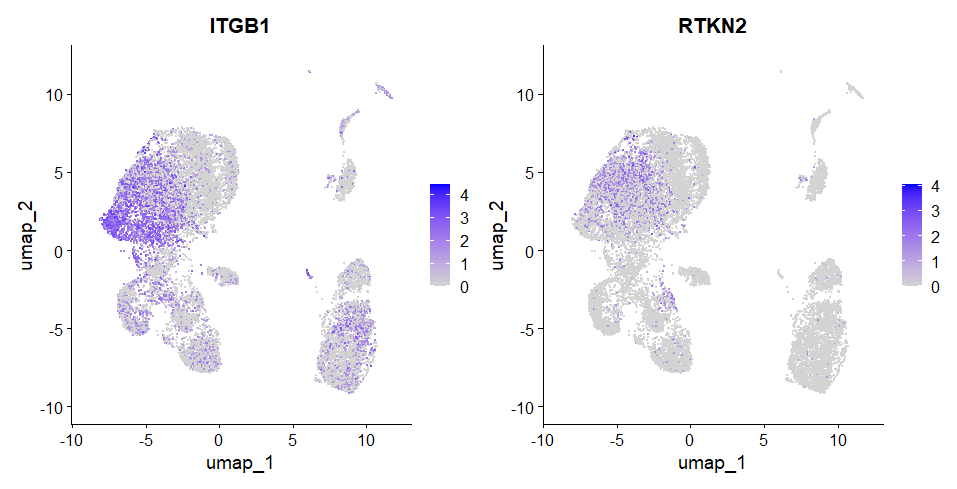
特征可视化也可以看出,ITGB1在整张图上都是有的,这就说明他的差异性并不是很显著
4. 细胞标注
这步很重要,并且这和聚类效果息息相关,在细胞标注中,分为singleR自动标注与手动标注两种,后者的精确值是最高的,所以如果没有特殊情况的话,强烈要求使用手动标注。
手动标注同时需要十分强大的生物知识(我不会QAQ)。具体来说步骤如下:
- 查看差异基因后,查询数据库和文献。文献先不提了,数据库可以使用这三个:Cellmarker,singleCellBase, HCA, MCA。数据库中包含人,鼠,猴等等,其中人的最多。
- 评估可以使用交叉验证,即使用多个标志基因组合进行注释并进行交叉对比
- 对于某一类的细胞群体,我们还可以手动为每个细胞群体赋予一个标签,如“CD4+ T细胞”、“中性粒细胞”等
tips:对于聚类来说,我们可以先对其进行一个大的聚类,将细胞大群先注释出来,随后在细化聚类
经验:log2FC的本质就是簇中对比差异性,越大就越说明与其他簇的分布情况不同,相对应的,这在找marker的时候也更方便,而p值只是一个假设检验的值。所以在寻找差异基因的筛选条件时,优先log2FC(云经验,哥们也没做过细胞标注)
需要代码直接联系本人哦,下方评论区or 邮箱:zhouqijia1110@gmail.com