质控与聚类
1. 测出数据部分
在通过前文的处理之后,我们得到了两个输出文件,分别为raw_feature_bc_matrix和filter_feature_bc_matrix。前者为原始数据,后者为cellranger经过自己处理后的数据,后续的分析会基于filter_feature_bc_matrix文件夹(上游比对分析产�生的三个文件)。文件夹目录如下
--filter_feature_bc_matrix
----barcodes.tsv
----features.tsv
----matrix.mtx
逐一解释:
**barcodes.tsv:**细胞标签
**features.tsv:**基因ID
**matrix.mtx:**表达数据
后续我们会使用seurat(R语言)进行分析
2. 质量控制
2.1 数据预处理
导入包的过程忽略了
首先我们先加载数据集,得到的数据应该如下所示:
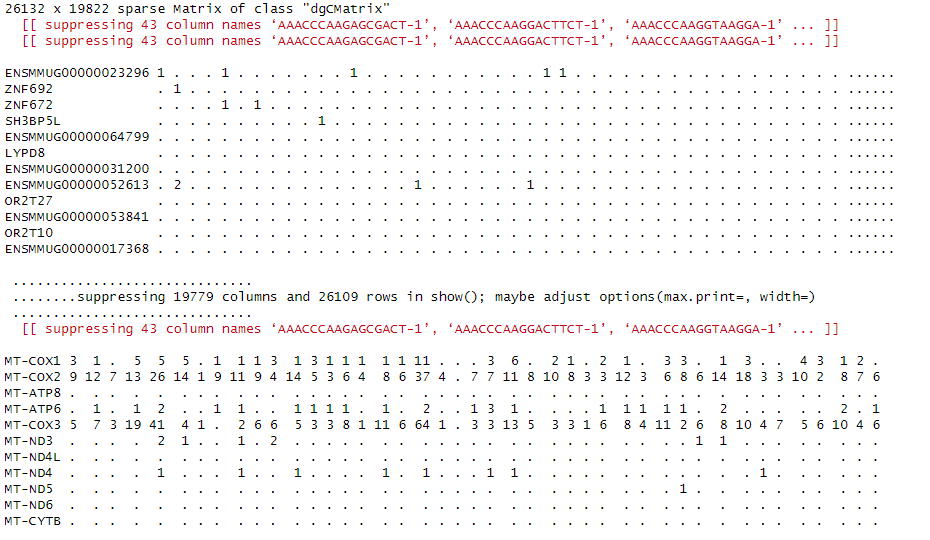
这是一个稀疏矩阵(这很重要,在后续分析中稀疏矩阵占用内存会比疏密矩阵小很多,不然在大数据集的情况下内存会爆炸);行代表基因(或者特征,取决于数据类型);列代表细胞。值代表特定细胞中某个基因的原始 UMI(Unique Molecular Identifier)计数。
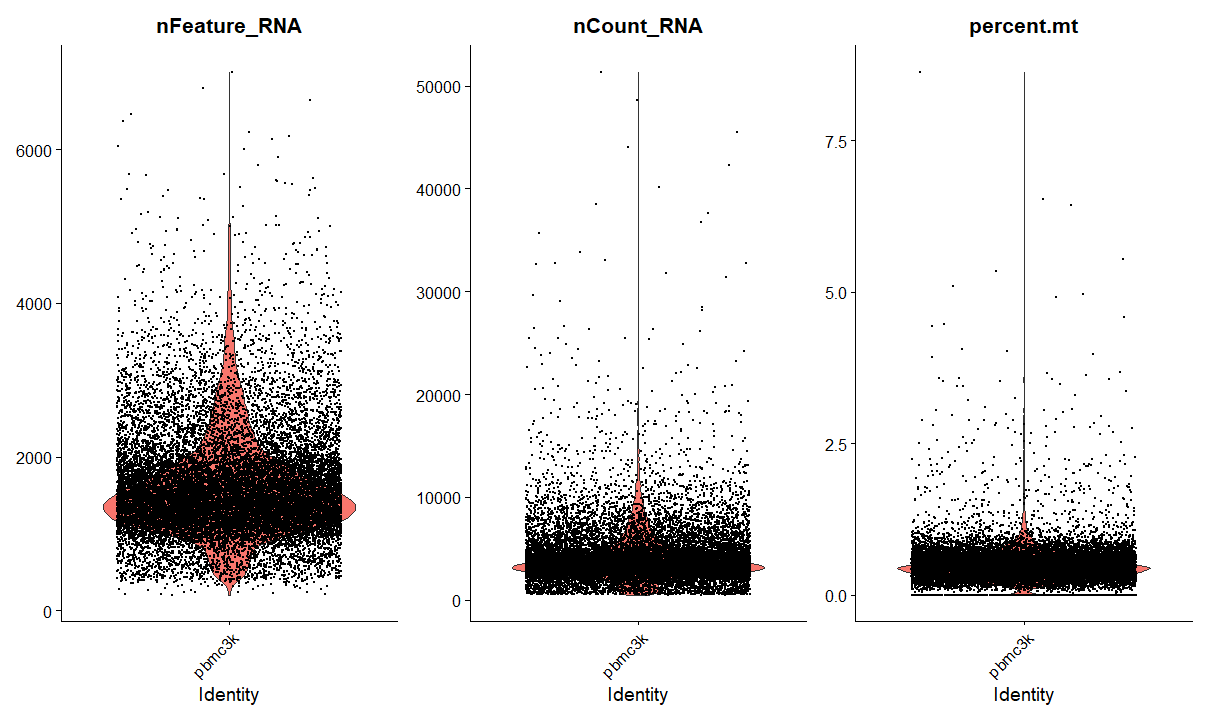
接着,我们需要先进行初步的筛选并查看数据分布,小提琴图与箱线图都是常用的办法,在此我们选择最为常用的小提琴图(更明了)。默认筛选条件为:
if(表达基因数<200 or 表达基因<3个细胞)
delete
可能来自一定比例的低质量细胞(比如细胞破碎造成细胞质RNA流失)。由于线粒体比单个的转录本大,不容易在破碎的细胞膜中漏出,从而导致测序结果显示线粒体基因的比例在细胞内占比过高。因此,质量控制这一步的目的就是把这些低质量的细胞去除掉。最后计算线粒体基因占所有基因比例后,做小提琴图
我们也可以绘制散点图,查看线粒体和基因数异常分布的数据点
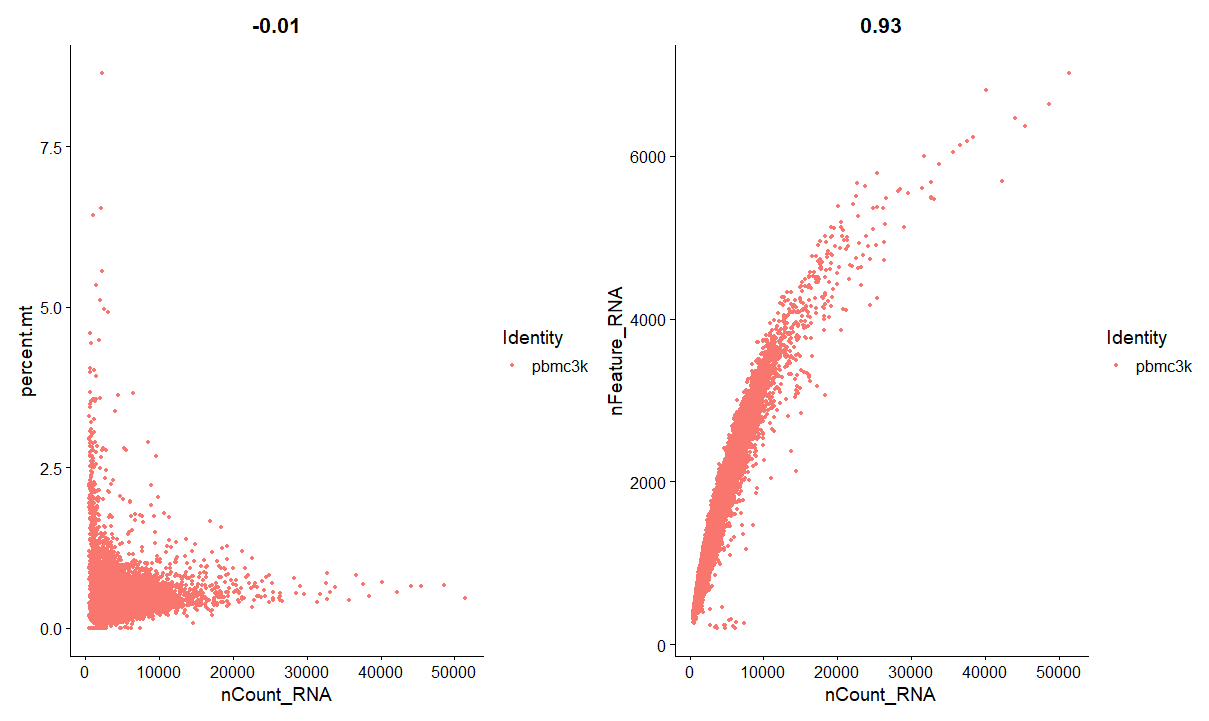
得到初步的分布情况之后,我们就需要根据可视化的情况进行筛选了,在此再进行条件过滤
if(表达基因数目<200 or 表达基因数目>5000)
delete
if(线粒体基因占比 > 20%)
delete
这里的阈值都需要自己做条件,请按照可视化后的结果自己调整,举个栗子,心肌细胞,本身就含有较多的线粒体,可以根据实际情况适当的调整阈值
高变异基因
这步的筛选可以理解为:如果有些基因的表达量都非常高并且每个细胞中表达水平十分相似,那就说明这一些基因的变异度很低,无用可以筛除。高变异基因的筛选方法有:基于方差(vst),基于离散程度,基于负二项分布,基于熵值等等。vst是较为常用的办法。在此默认我们保留2000个基因,得到结果如下
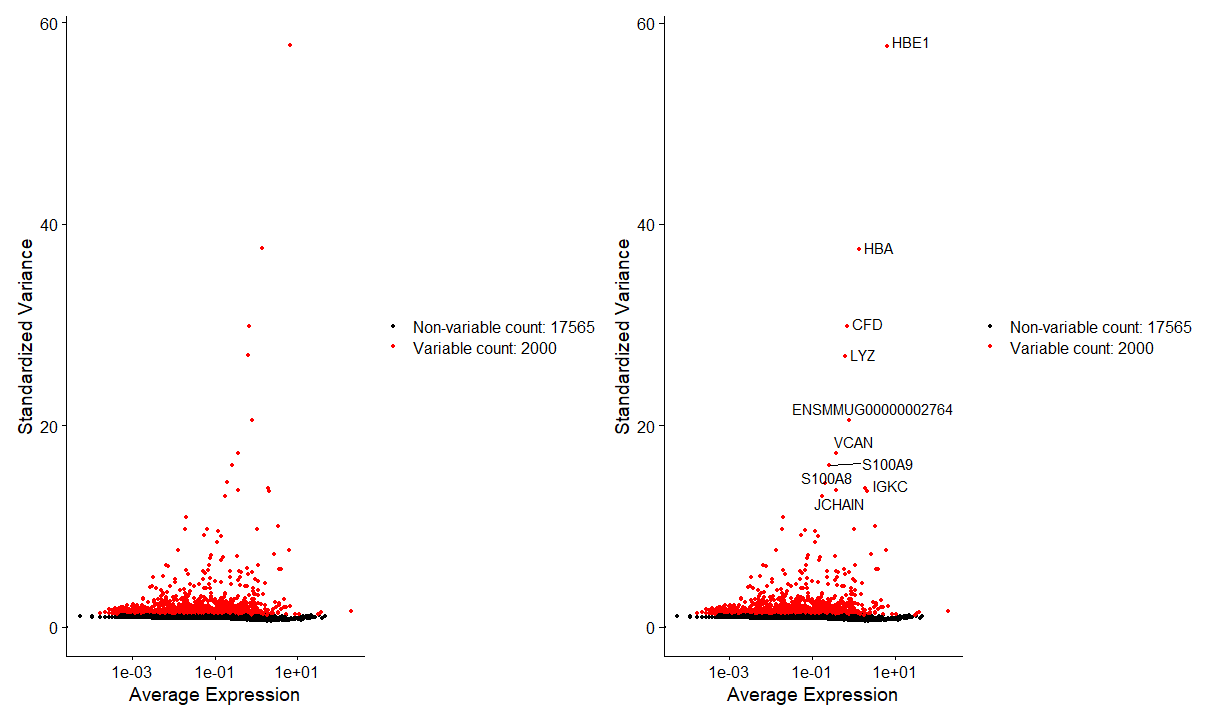
可以看到,我们对标准化与未标准化的数据都进行了比较,标准化后的数据表现更好,使用log1p,即表达量的对数加1;使用其他办法如标准化到10000也是可以的
2. 聚类分析
2.1 降维
在进行聚类之前,我们需要首先对数据进行降维,降维之后可以将很多不必要的特征删除。PCA是最为常用的办法,我们可以通过前文筛选出的2000个变量进行降维
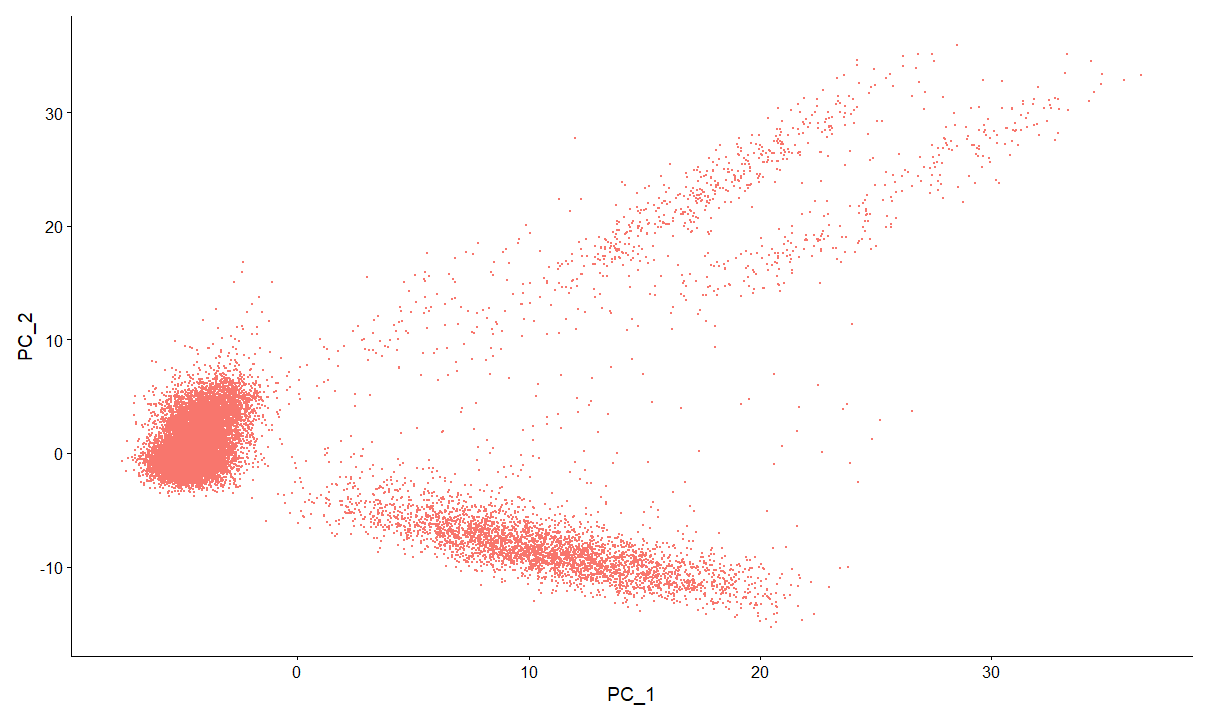
在降维之后,我们可以查看PC_1与PC_2中单个细胞之间表现出相关性

也可以查看在两个维度中,数据的分布情况。以主成分PC1、PC2为例,展示主成分分析得到的细胞分布图。
最后画出碎石图,这直接决定了后续我们聚类所采用的参数
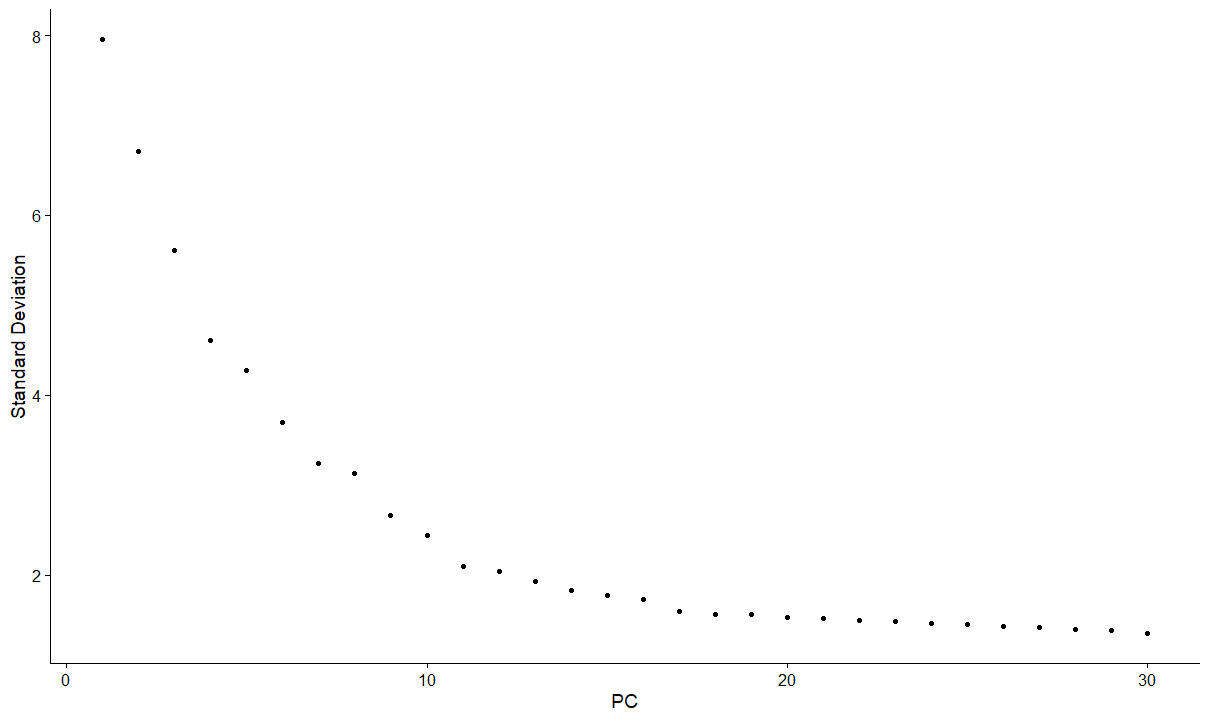
如何选择参数?
很简单,寻找拐点位置,并且这个拐点(比如说dim=20)的解释量必须要大于90%。
for dim in range(50):
if dim_for_exp > 90%:
break
tips:有人在论文中做过实验,一般我们取30左右进行,其实25-35都不会很影响后续的分析,但这个必须按照自己画出来的碎石图进行判断
当然,也可以通过Jackstraw分析判断选取成分数量
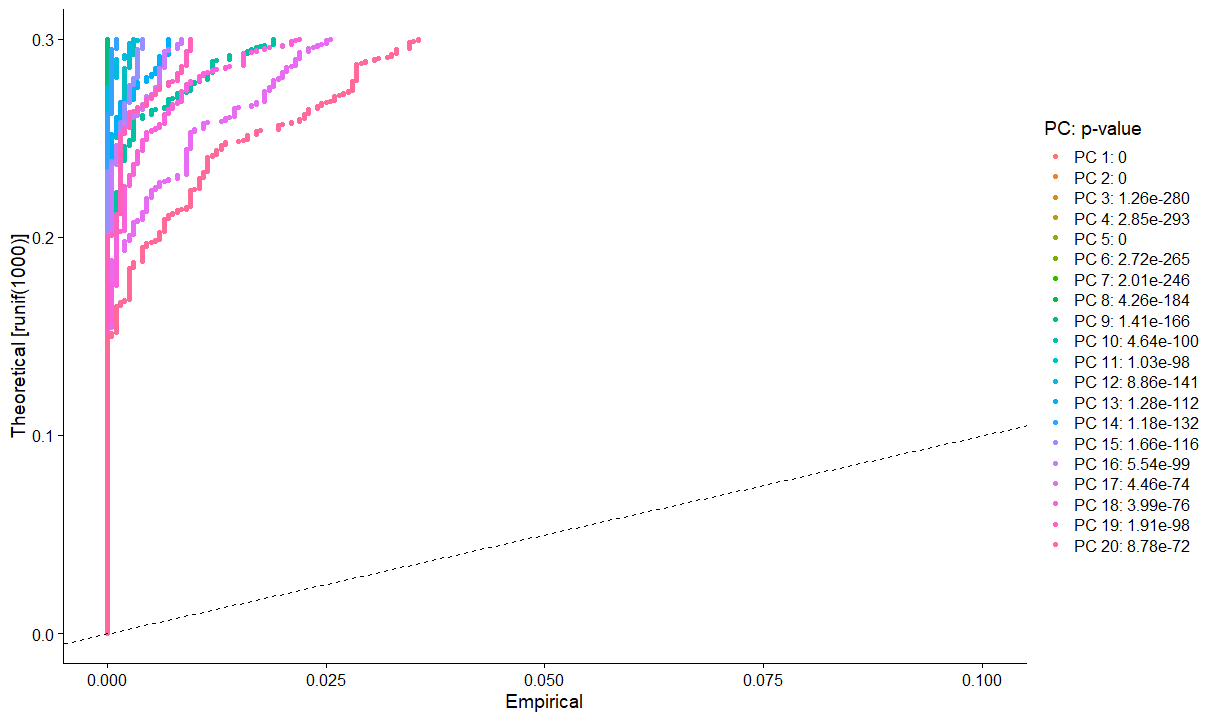
只要高于虚线,dim都是可取的,本文由于电脑配置原因跑起来太慢了,因此只画了20个==
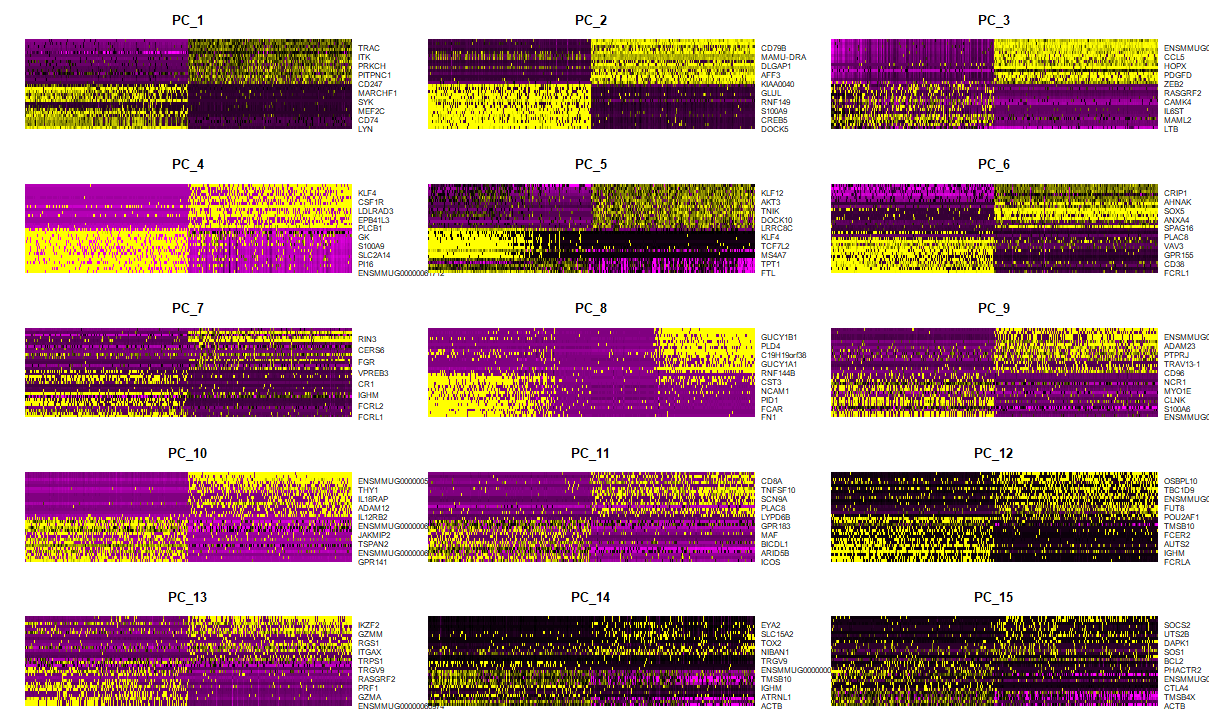
也可以绘制维度热力图,探索数据集中异质性的主要来源(主打一个图多好看)
2.2 聚类
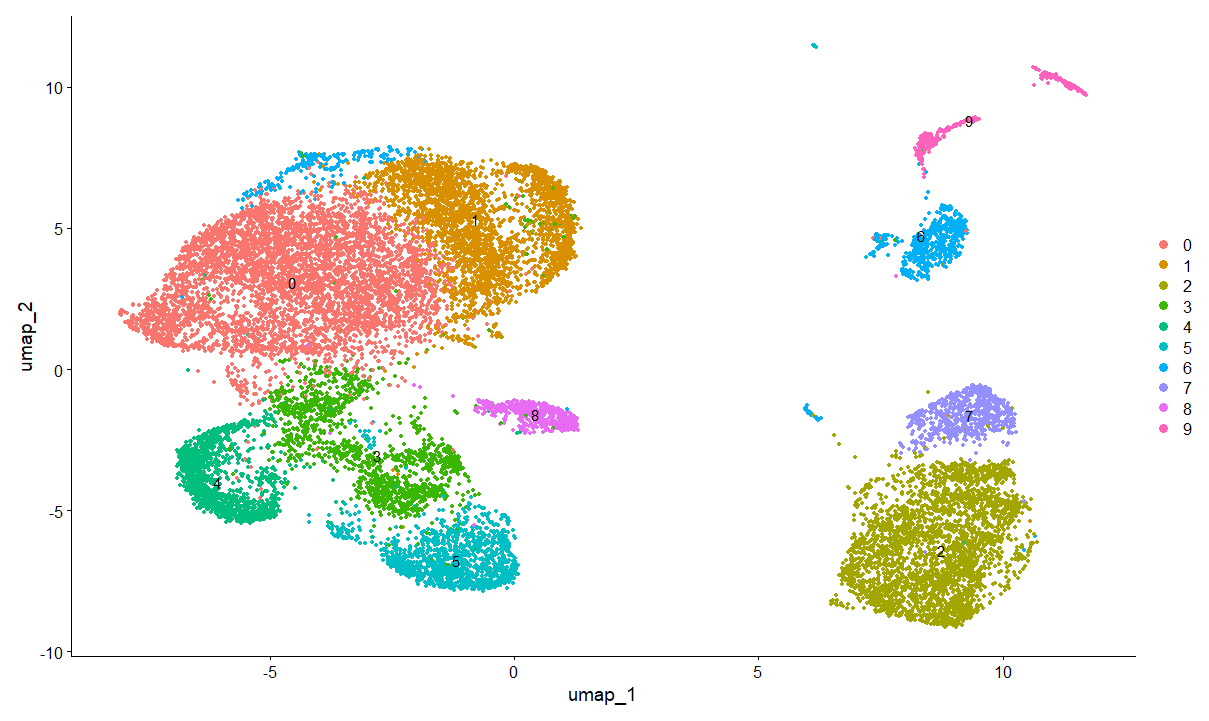
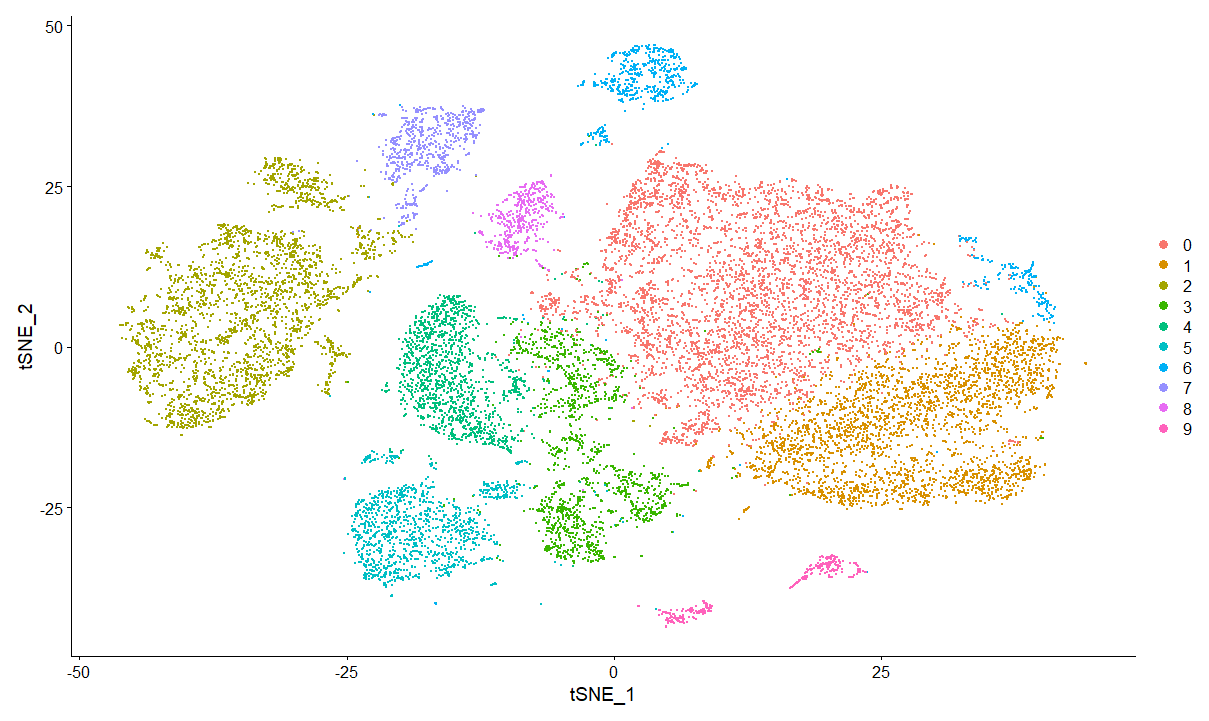
目前,主流的聚类办法有两种,分别为umap和t-sne。前者聚的更紧凑,后者
Umap聚类效果更为紧凑,簇间近且边缘轮廓也较为靠近
T-sne相反
你以为这就结束了?大错特错!聚类有很多重要参数,不同的参数聚出来的效果是截然不同的。接下来依次说明:
Neighbors:单个细胞认为自己邻居数量的半径,这个参数可以由上文的pca得出
ClustersNumber:簇数量,可以通过leiden算法得出,其中的resolution也需要自己手动控制
理论上来说,簇数量是由真实世界决定的,比如说表皮细胞,有多少个就聚多少个簇,上文的所有办法都是为这个服务
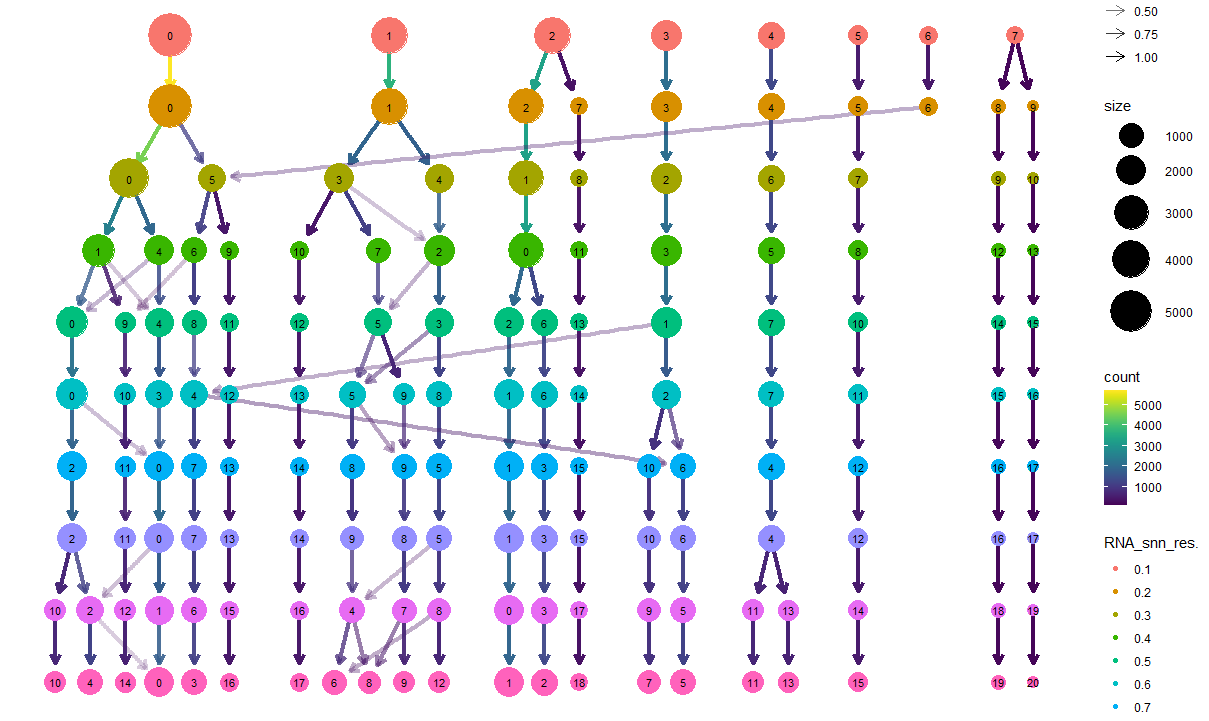
当然,在前期我们不知道数量的时候,可以通过决策树来决定resolution的值到底是多少,以次先做一个大致的观察。原则是不要交叉,交叉就暂停。整一张抽象的图,按照这样的话就取0.2
需要代码直接联系本人哦,下方评论区or 邮箱:zhouqijia1110@gmail.com