富集分析
通过前文我们可以提取出差异基因,然而差异基因的数量较多,对其进行分析会十分冗长,因此我们可以采取富集分析的方式进行归类。富集的意思是表示差异基因或者差异物质中注释到某个代谢通路的基因或者物质数目在所有差异基因或者物质中的比例显著大于背景基因或物质中注释到某个代谢通路的基因或物质数目在所有背景基因或者物质中的比例。简而言之一句话概括:该差异基因在特定的通路上占比很大
5.1 GO富集分析
主要用来看基因的三个方面,分别是分子功能、细胞组分、参与的生物过程。
举例,铁离子结合的GO term是GO:0005506,如果我们对所得到的差异基因进行GO富集分析后得到该term富集,则我们可以认为我们所研究的现象可能与铁离子结合有关系
进行go分析时,可以得到如下数据:
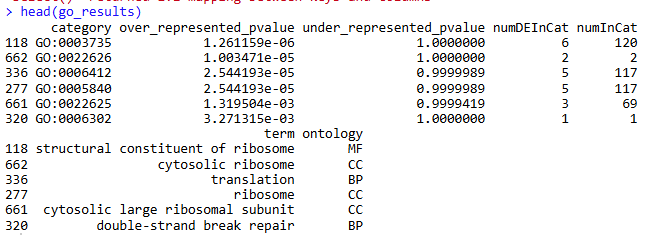
参数解释:
category: Gene Ontology数据库中唯一的标号信息
**over_represented_pvalue:**富集分析P值,P 值越小越显著
under_represented_pvalue:
**numDEInCat:**该功能类下的差异基因数目
**numInCat:**该功能类下的基因数目
**term:**Gene Ontology功能的描述信息
**ontology:**该GO的类别(CC,细胞组分;BP,生物进程;MF,分子功能)。
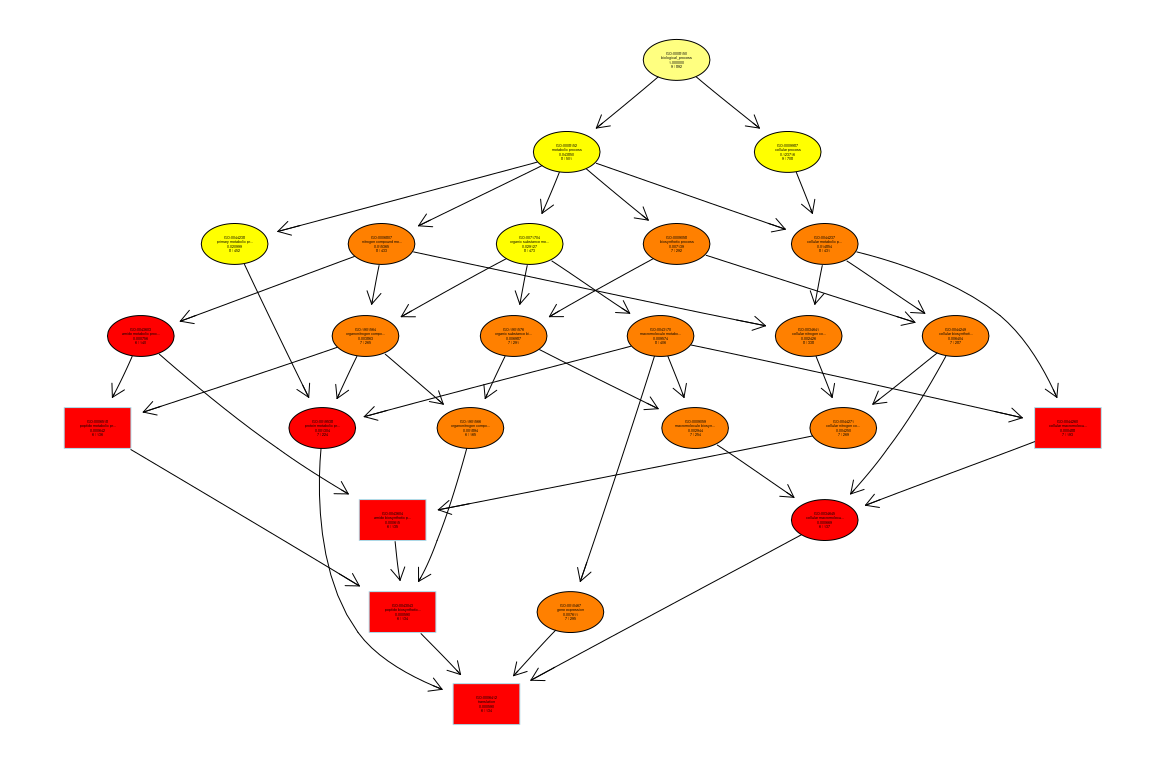
接着,还可以可视化DAG图,分支代表包含关系,从上至下所定义的功能范围越来越小,一般选取GO富集分析的结果前5位作为有向无环图的主节点,颜色的深浅代表富集程度。概括的说, 可以分析GO terms在富集分析中是否显著,并且terms是如何相互关联的
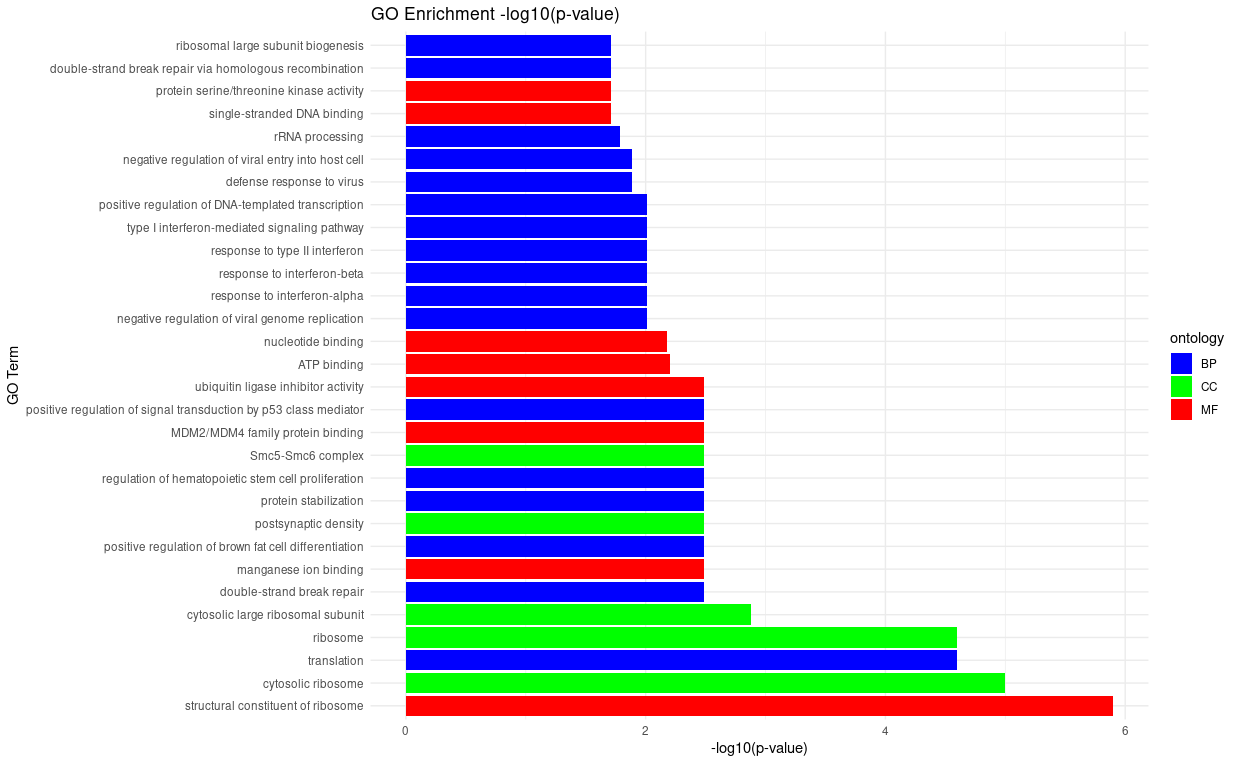
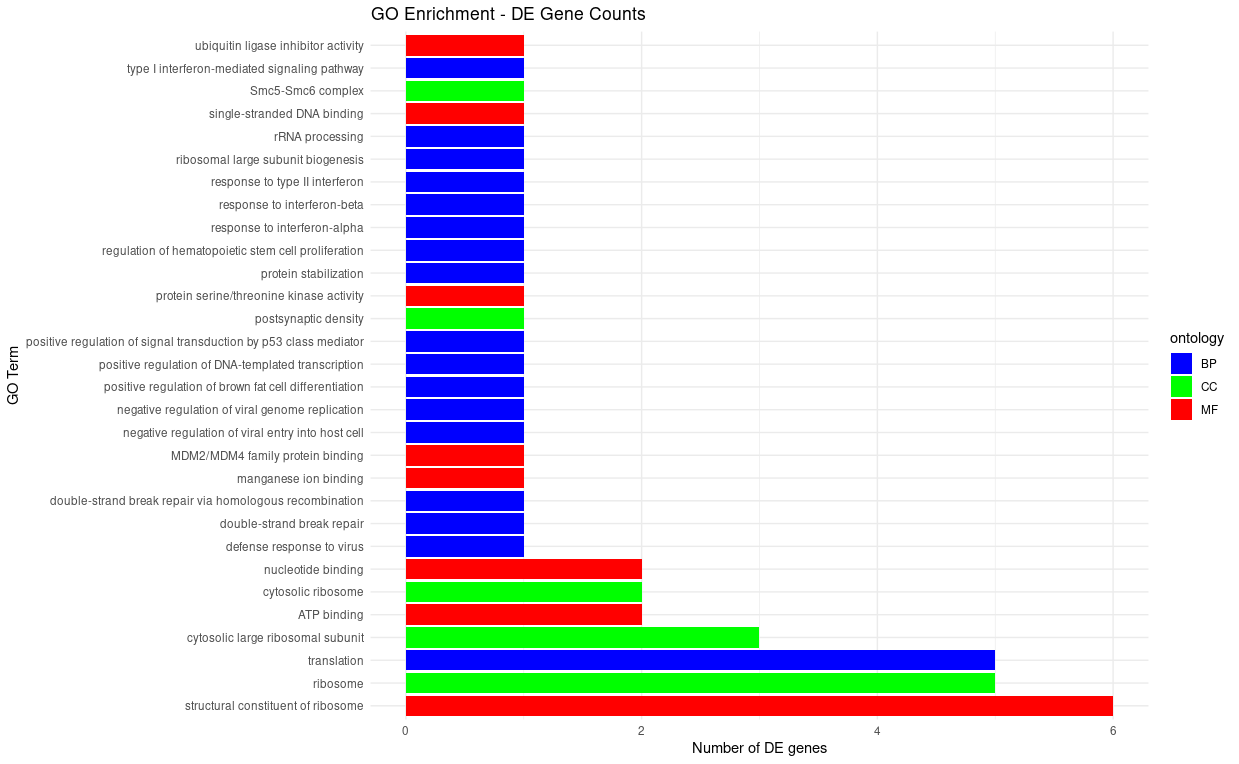
最后,也可以通过柱状图将ontology进行分类,以柱状图呈现出来
5.2 KEGG分析
在生物体内,不同基因相互协调行使其生物学功能,通过Pathway显著性富集能确定差异表达基因参与的最主要生化代谢途径和信号转导途径。KEGG(Kyoto Encyclopedia of Genes and Genomes)是有关Pathway的主要公共数据库(Kanehisa,2008)。Pathway显著性富集分析以KEGG Pathway为单位,应用超几何检验,找出与整个基因组背景相比,在差异表达基因中显著性富集的Pathway
通过KEGG分析后,我们可以得出如下表格(注意:使用enrichKEGG后传入的是一个对象,建议转为数据框,方便后续的作图与分析)
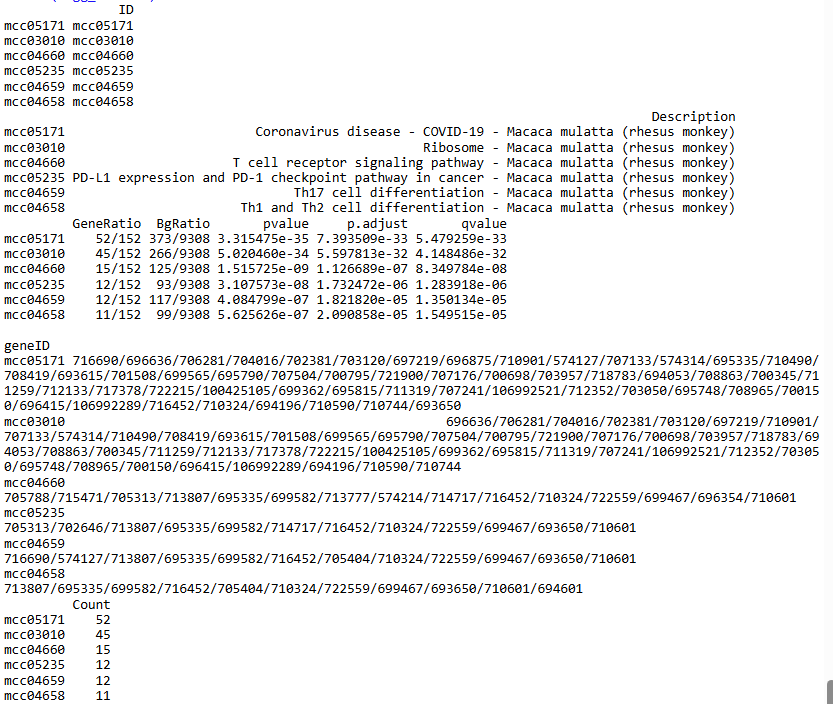
表格有些大,主要是geneID太长了,分别解释各字段含义:
**ID:**KEGG的PATHWAY数据库中途径标识
**Description:**该通路的描述
**GeneRAatio:**富集到该通路里的差异基因数/全部可以富集到KEGG里的差异基因数,比例越高说明越富集
**BgRatio:**该通路的全部基因数/该物种全部有KEGG信息的基因数
**pvalue:**p值,不过多解释了
**p.adjust:**矫校正过的p值
**qvalue:**q值
**geneID:**富集到该通路里的基因的名称
**Count:**富集到该通路中的差异基因的数目
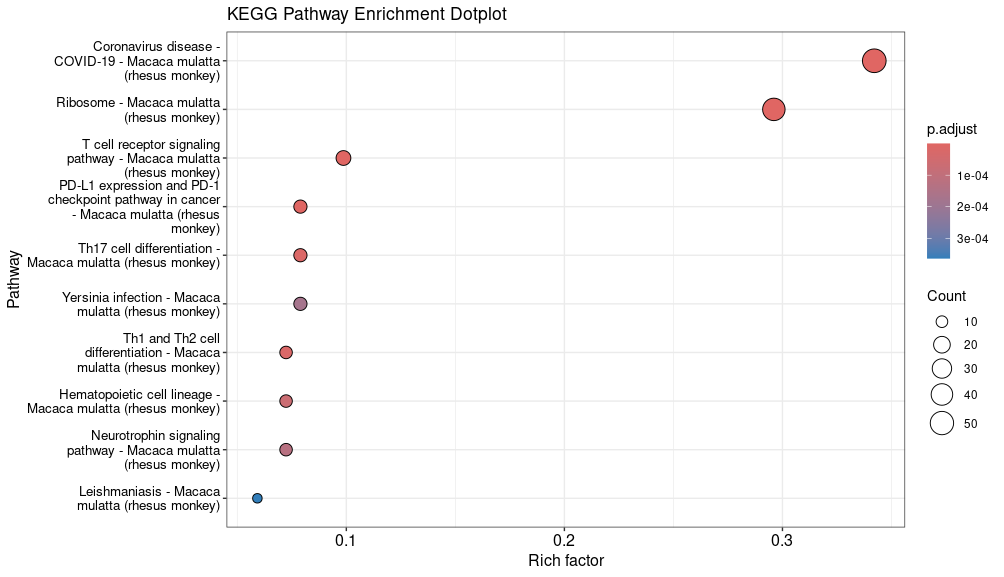
接着,我们就可以画出点图与柱状图了。先说前者,这个只需要一行代码就行
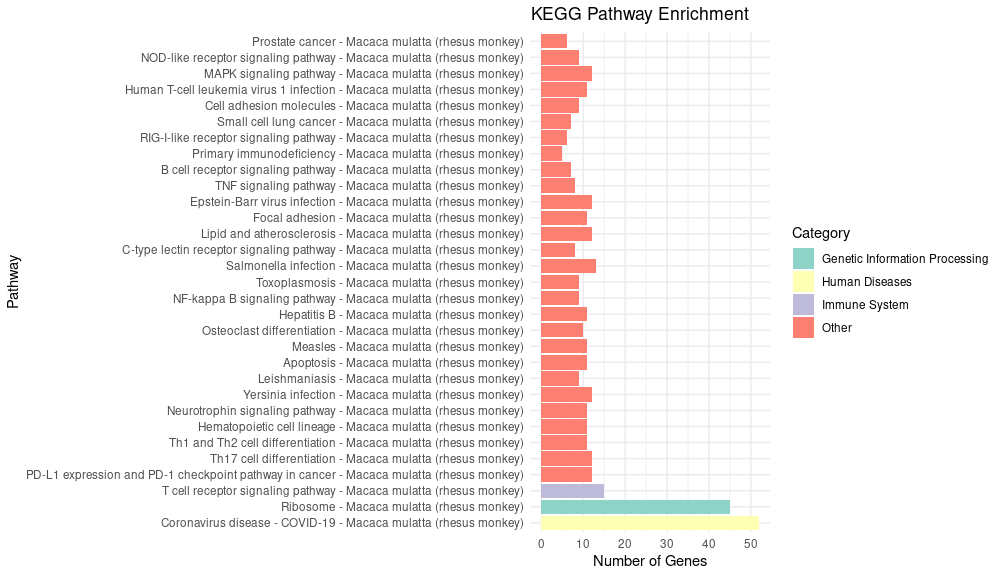
在绘制柱状图的时候,同时还需要注意柱子颜色的分类。在其他资料中观察到�他是按照通路类别进行分类的,一共有如下七种:
- Metabolism(代谢)
- Genetic Information Processing(遗传信息处理)
- Environmental Information Processing(环境信息处理)
- Cellular Processes(细胞过程)
- Organismal Systems(有机体系统)
- Human Diseases(人类疾病)
- Drug Development(药物开发)
我们可以通过KEGG官网,以id为key进行辨别
- 登录KEGG PATHWAY Database (genome.jp)
- 输入id
- 查看类型后手动标注
以两个我瞎编的为例,柱状图最后应该长这样
批次效应
用于多样本整合时才需要,不过多赘述(因为我没有)
5.3 Reactome
这个其实和前面两个是一样的,区别仅仅是换了一个数据库
# 使用ReactomePA进行通路富集分析(只有human)
reactome_results <- enrichPathway(gene = entrez_genes,
organism = "human",
pvalueCutoff = 0.05,
readable = TRUE)
我是做的猴子,这个数据库听说只有人鼠,具体还有什么可以自己百度一下
5.4 GSEA
这块内容其实和前文的GO与KEGG分析相同,主要不同在于GO富集分析是先筛选差异基因,再判断差异基因在哪些注释的通路存在富集;这涉及到阈值的设定,存在一定主观性并且只能用于表达变化较大的基因,即我们定义的显著差异基因。GSEA则不局限于差异基因,从基因集的富集角度出发,理论上更容易囊括细微但协调性的变化对生物通路的影响。
图表与上文是一样的,不过多赘述