拟时序分析
在进行了聚类之后,其实各细胞是否具有同种生存状态是未知的。拟时序分析的目的就在于将细胞分为不同的分支,将各点(细胞)体现在不同的时间坐标中,从而了解各细胞的状态定位
在做拟时序分析的时候,采取的是机器学习方法(无监督和有监督),因此需要一定的生物学知识对图标进行判断,图中主要是为了表达细胞之间(簇)表达谱系的连续性,因此方向未必与现实情况相同(需要在代码中加入reserve)
举个例子:B细胞不会分化为NK细胞,但在图中就会如此,这就是reserve的作用
本文主要采取无监督的方法进行分析
tips:无监督就是没有真实数据,有监督就是包含一定的真实数据
- 无监督数据:常见的如单细胞 RNA 测序数据,在特定的发育阶段采集了样本,但不确定细胞的确切时间顺序。
- 有监督数据:例如药物处理实验,在不同时间点采集了单细胞样本,记录了每个样本的处理时间,通过这些时间点信息可以进行有监督的拟时序分析。
首先,使用monocol2创建CellDataset对象后,就有了拟时分析结果的可视化,我们可以将其分为:
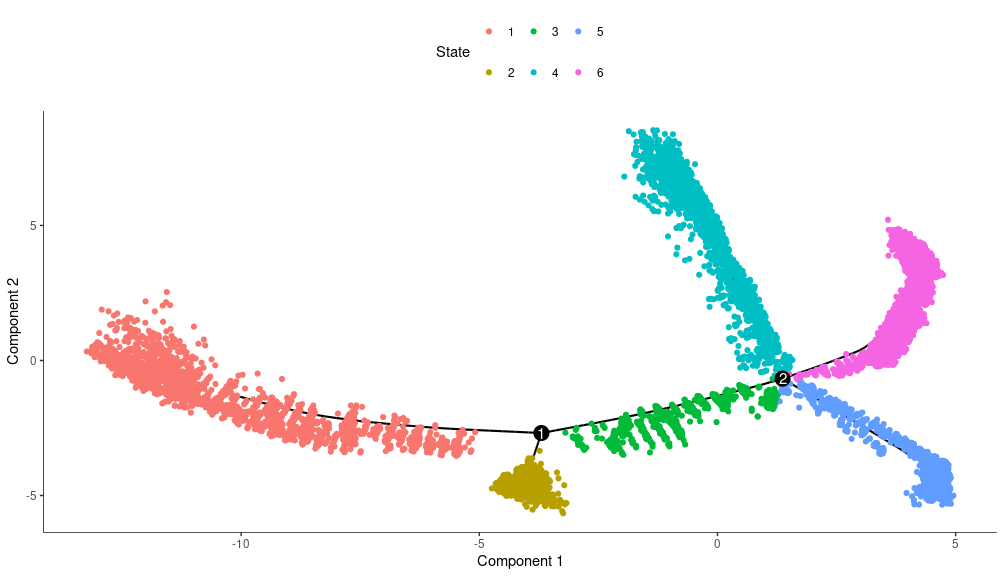
state状态:
代表了细胞在某一生物学过程中所处的不同阶段。例如,在细胞分化过程中,初始的未分化状态、不同分化路径中的中间状态,以及终末分化状态,都会被标记为不同的“state”。
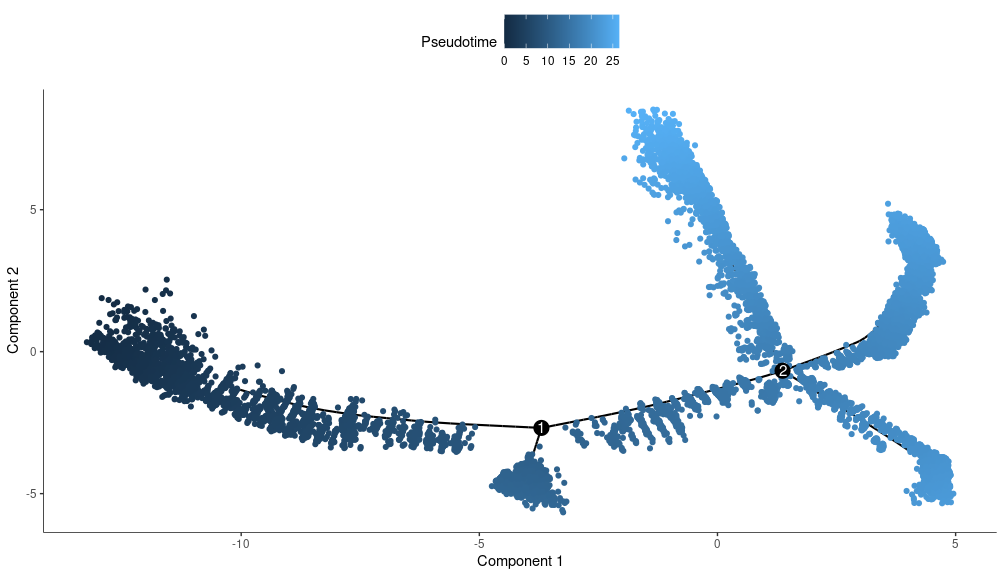
Pseudotime时间:
这是算法模拟出来的一个时间轴,越接近0就说明越早
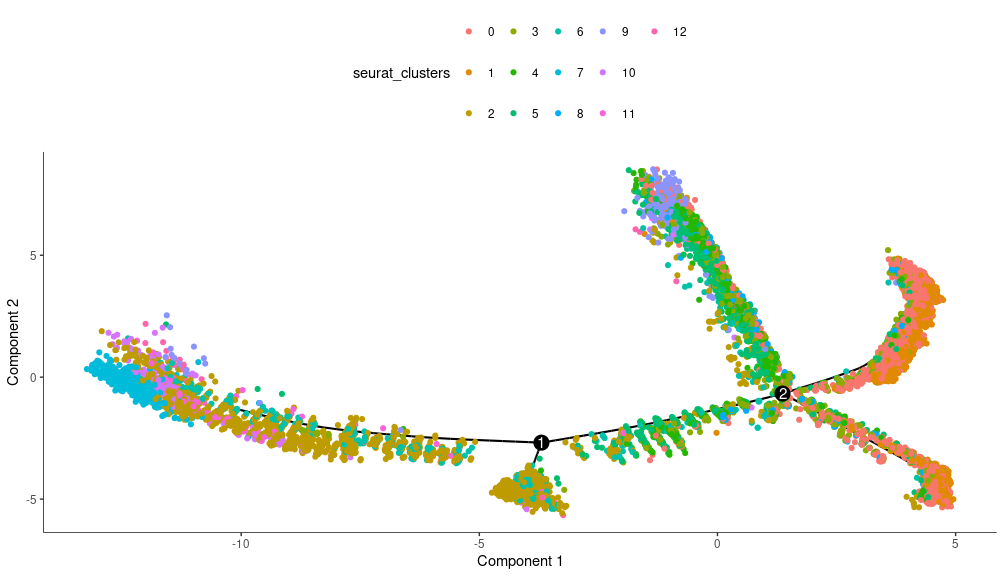
Seurat分群结果:
这其实就是之前用umap聚出来的为基准了,按其基因表达模式进行分类
细胞注释结果(前文没有注释,如果想要的话可以直接color_by=''labels'')
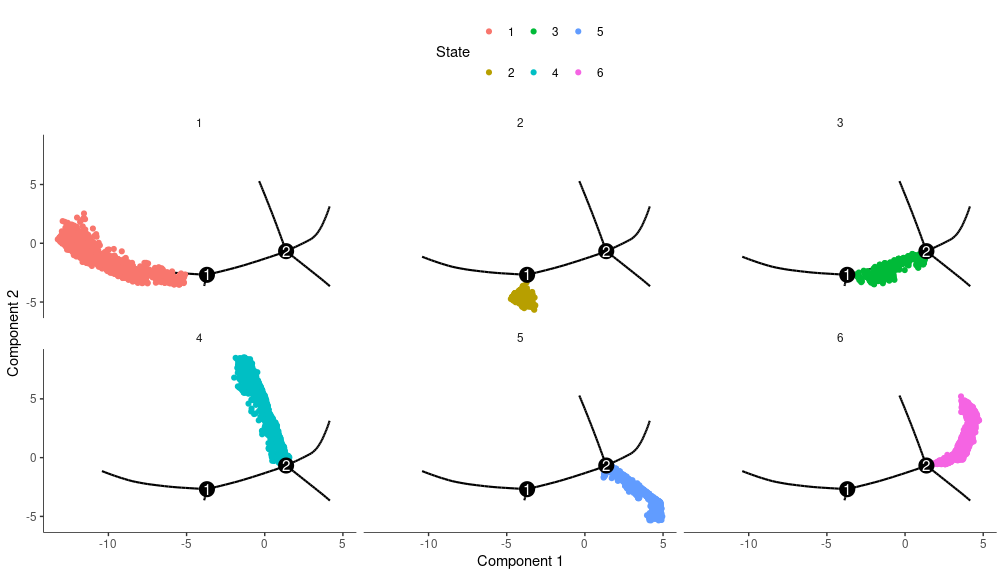
state状态分支()
其实就是讲state状态分开来,更直观的看出各类处在什么分支上
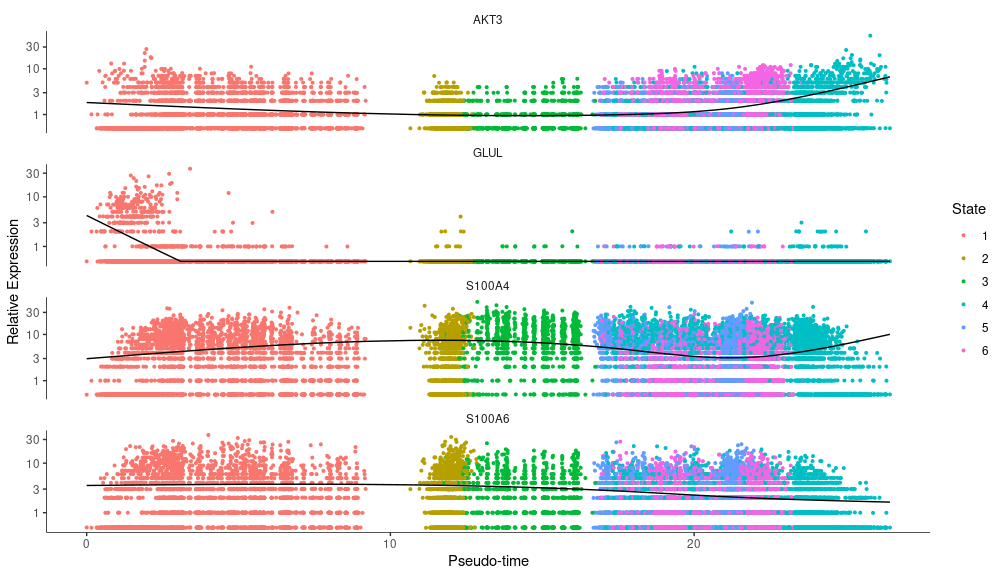
在筛选完差异基因后,可以观察单个差异基因的表达情况。选择差异基因的原因的在整个分化过程中更加关键,能够揭示不同状态或分支间的生物学差异。
横轴代表拟时间,通常用来模拟细胞从一个初始状态到最终状态的进程;纵轴代表基因表达量,上面的位置表示在特定拟时间点上,基因的表达水平。
分支点设定在1的时候,表示关注在第一个分支点处的基因表达变化,细胞分类为3说明分为三个不同的簇。颜色通常表示基因的表达水平,颜色越深,表达越高;颜色越浅,表达越低。热图通过颜色变化来展示基因在不同细胞分支中的表达情况
这个热图用于展示在细胞分化过程中,不同分支上的基因表达模式。通过观察基因在分支上的表达差异,可以获得这些基因在不同细胞命运中的潜在角色,即哪些基因是上调的,哪些是下调的,从而反映基因表达的动态变化。
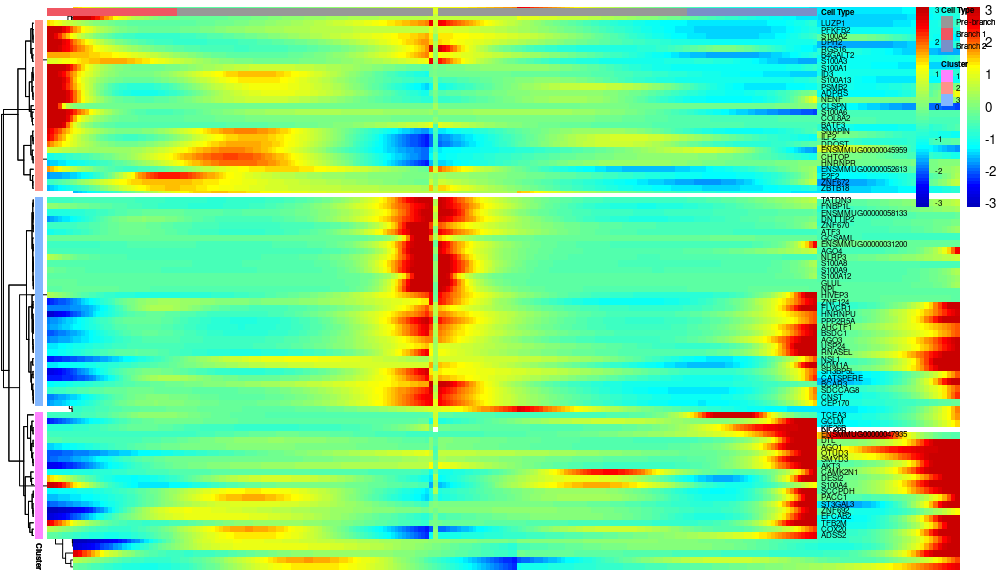
(图片分辨率或许有一些问题,自己画的时候可以调整一下width和height)